La nueva epidemia: los pepinos, presuntos culpables
Miguel Ángel Cevallos y José Luis Puente

Foto: Arturo Orta
Un brote epidémico surgido en Alemania hace unos meses alertó a las autoridades mundiales de salud. Ésta es la historia de cómo se identificó a la bacteria responsable, gracias a los avances en ciencias genómicas.
Como microbiólogos, siempre nos han llamado la atención las epidemias; en especial saber cómo surgen, cuáles microorganismos las causan y qué efectos tienen en la salud y en otras facetas de la actividad humana. Además no deja de maravillarnos la capacidad que tiene hoy la ciencia para reconocer con prontitud los brotes epidémicos, aislar e identificar a los organismos involucrados en ellos y atajar, o al menos controlar, sus avances.
Sirva como ejemplo la epidemia que hace poco alarmó a las autoridades mundiales en salud pública: en los primeros días de mayo de este año, en Alemania se detectó un número inusualmente grande de pacientes con síndrome urémico hemolítico, enfermedad frecuentemente provocada por una variedad de bacteria patógena que se conoce como Escherichia coli enterohemorrágica O157:H7 o, para facilitarnos la vida a todos, EHEC O157:H7.
Los individuos que, por haber consumido agua o alimentos contaminados, se infectan con esa bacteria, sufren de fiebre, dolor abdominal y diarreas sanguinolentas graves. Sin embargo, ese padecimiento todavía puede ser peor: a veces, algunos desventurados desarrollan complicaciones secundarias como el síndrome urémico hemolítico, que se caracteriza por fallas renales serias y por la disminución drástica del número de glóbulos rojos, situación que, aun con los cuidados médicos pertinentes, puede arrastrarlos a la sepultura.
Ante el incremento súbito del número de pacientes con dicho síndrome —en pocos días se acumularon cientos de casos—, el Instituto Robert Koch de Alemania inició una investigación exhaustiva para identificar dónde surgieron las infecciones y a su agente causal. También estimó prudente —sin tener aún todos los datos en la mano— aconsejar a su población no consumir pepinos y otras verduras de las cuales inicialmente se sospechó que podían estar contaminadas con la bacteria patógena. Dada esta advertencia, Alemania cesó de importar hortalizas de España, principal país abastecedor de su mercado, causando así a los agricultores ibéricos un grave perjuicio, que se acentuó dramáticamente cuando otros países también vetaron la importación de dichos productos. Este aviso prematuro del instituto germano les costó a los españoles literalmente millones de euros, pues, amén de provocar que de dos plumazos, por así decir, los agricultores se quedaran con sus productos en el almacén, dio origen a lo que se conoce hoy como la crisis de los pepinos.
Desde luego, el problema económico no era el único factor a tomar en consideración en esta crisis: de mayor trascendencia, como veremos, era el que se refería a la salud mundial.
Ante la seriedad del brote epidémico, los alemanes dieron aviso al Centro Europeo para la Prevención y el Control de Enfermedades, cuya sede se halla en Estocolmo, y solicitaron su intervención. Con esta acción se inició una carrera contra el tiempo para descubrir la verdadera fuente de la contaminación e identificar, estudiar, y controlar la bacteria responsable de esta crisis de salud.
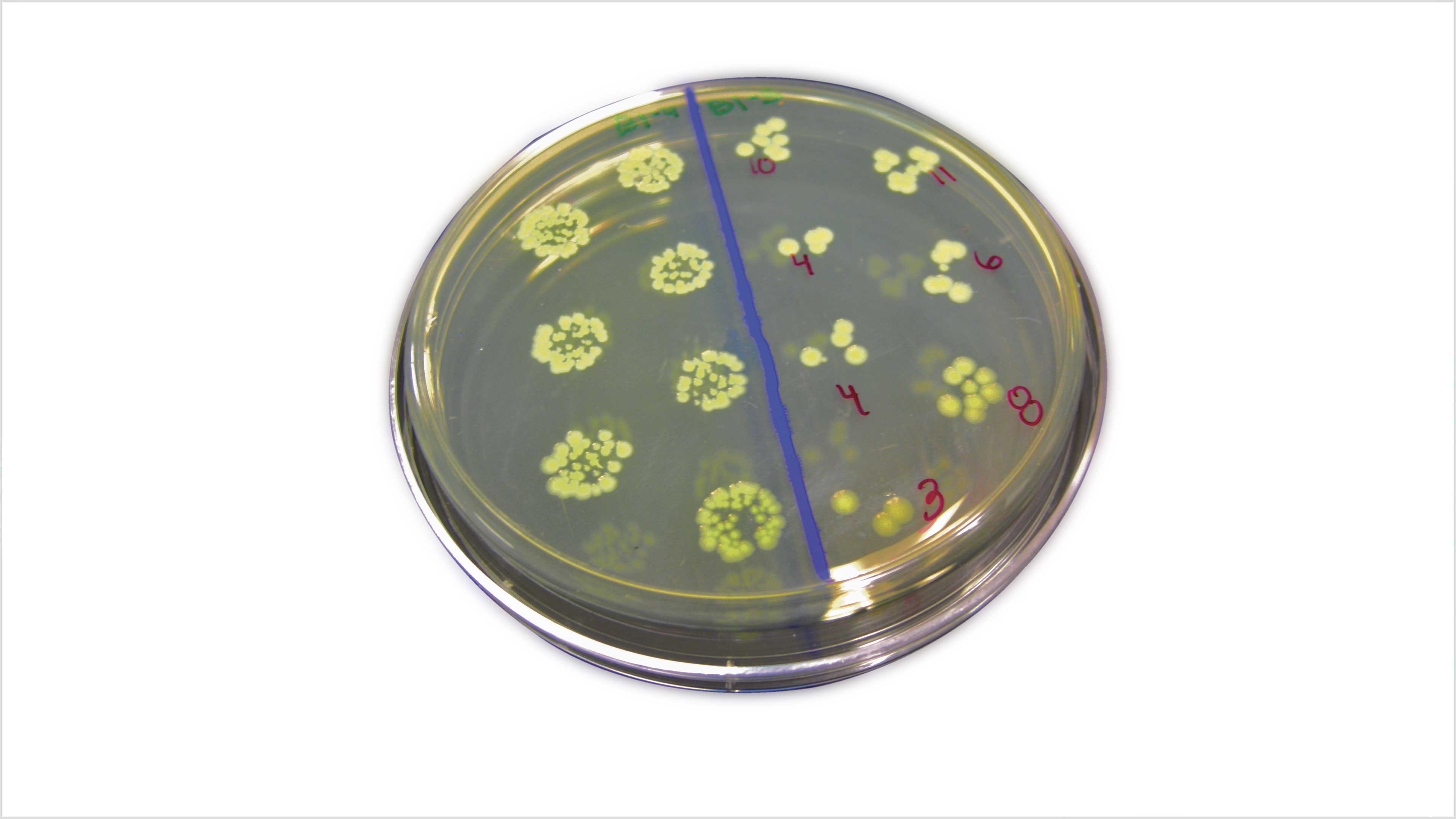 Cultivo de E. coli en caja de Petri. Foto: cortesía NASA/Ames.
Cultivo de E. coli en caja de Petri. Foto: cortesía NASA/Ames.
Germinados en el banquillo
Los epidemiólogos europeos, tras una muy detallada investigación de los casos, descubrieron que muy posiblemente los pacientes se contagiaron por haber consumido germinados de diversos vegetales en algunos restaurantes de la ciudad alemana de Lubeca. Estos restaurantes obtenían sus germinados de compañías agrícolas de la ciudad de Uelzen, situada en el norte de Alemania. Una vez identificada la fuente de la epidemia (ver recuadro), las autoridades de salud germanas tomaron medidas para controlarla y evitaron que siguiera avanzando. De hecho, desde los primeros días de agosto había claras evidencias de que la epidemia en el país teutón estaba totalmente bajo control. Para que apreciemos la rapidez con que se expanden las epidemias en el mundo moderno, apenas tres meses después del inicio de la epidemia de EHEC, se enfermaron 4 075 personas de 16 países, de las cuales 50 fallecieron. La mayor parte de los pacientes no germanos se contagiaron porque hicieron turismo en Alemania y tuvieron el mal tino de comer algún alimento con germinados contaminados.
Inesperadamente, a finales de junio un nuevo brote de esta epidemia se detectó en Burdeos, puerto del sudoeste de Francia, y los afectados (ocho) no habían visitado Alemania, ni estado en contacto con turistas que hubieran visitado recientemente este país. Algunas evidencias preliminares indican como fuente inicial de ambos brotes epidémicos a semillas de fenogreco, originarias de Egipto, que se utiliza como condimento y planta medicinal, y últimamente en la producción de germinados. Esas semillas habrían entrado al mercado europeo sin los controles sanitarios apropiados. Y si bien no se descartaron otras posibles fuentes secundarias, esta información permitió segar desde su origen este nuevo brote epidémico.
Asesino al descubierto
La Escherichia coli podría ser la protagonista de la famosa novela El extraño caso del Dr. Jekyll y Mr. Hyde del escritor escocés Robert Louis Stevenson, la historia fantástica de un hombre normal que se transforma en un malvado cada vez que bebe una pócima. La E. coli no patógena puede vivir inadvertida en nuestros intestinos, sin causar daño, o convertirse en un bicho maligno (E. coli patógena). Ambos tipos se pueden encontrar en los intestinos del hombre y de otros animales de sangre caliente y, por lo tanto, en las aguas negras donde su presencia sirve como marcador de contaminación fecal.
Los microbiólogos pueden distinguir en el laboratorio diferentes subtipos (formalmente denominados serotipos) de E. coli patógenas y no patógenas, con base en ciertas moléculas de la superficie de la bacteria —antígenos O, de los cuales existen 167 variedades— o del aparato con que nadan, el flagelo —antígenos H, de los que se reconocen 75—. Así, cuando a una Escherichia se le da la designación de E. coli O157:H7, significa que tiene en su superficie el antígeno tipo O157 y en su flagelo el H7.
El esquema de clasificación de las E. coli en serotipos, que se basa esencialmente en el trabajo que el investigador danés Fritz Kauffman publicó en 1947, permite hacer una cierta distinción entre las no patógenas y las patógenas, o evaluar su grado de peligrosidad como patógenas de acuerdo a cómo se asocian con brotes epidémicos previos.
Los primeros análisis de laboratorio indicaron que la bacteria responsable del brote epidémico que se detectó primero en Alemania era una variedad de E. coli patógena, inusual en todos sentidos: primero, se determinó que esta bacteria pertenece al subtipo O104:H4, que nunca había sido causante de brotes epidémicos de ninguna clase, aunque ciertos epidemiólogos aseguran que ya se habían aislado bacterias parecidas en Alemania (2001), Francia (2004), Corea (2005) y la República de Georgia (2009). Otra característica que llamó la atención de los investigadores es que la nueva bacteria produce una potentísima toxina (conocida como verotoxina o toxina tipo Shiga) que daña gravemente la pared interna de las arterias y de las venas de poco calibre del tracto digestivo, de los riñones y de los pulmones; por tanto, es la responsable de una gran parte de los síntomas que caracterizan al síndrome urémico hemolítico.
Una vez que se tuvo clara la identidad del enemigo, dos instituciones, una alemana, el Centro Médico Universitario de Hamburgo-Eppendorf, y otra china, el Instituto Genómico de Beijing (BGI), secuenciaron el material genético (genoma) de la bacteria en sólo tres días. El análisis comparativo del genoma mostró que el pariente más cercano de este organismo en realidad es una E. coli enteroagregativa (EAEC) que hace pocos años se aisló estudiando a pacientes africanos infectados con VIH (el virus que causa el sida) que sufrían diarreas persistentes. Éste y otros estudios de genómica comparativa mostraron que la bacteria causante del brote epidémico posee la capacidad de producir verotoxina, así como una combinación de genes que participan en la virulencia, que son típicos de las E. coli enteroagregativas. Por ello la bacteria nueva se ha clasificado como E. coli (EAEC VTEC) O104:H4 (las siglas VTEC significan que es una E. coli (EC) que produce verotoxina (VT). Para los propósitos de este artículo, a esta bacteria la denominaremos simplemente E. coli alemana.
Labor detectivesca
Para identificar el origen y la causa de una epidemia como la que surgió en Alemania en mayo pasado, se determina primero si todos los pacientes han sido afectados por el mismo organismo. Después se investiga si los casos están relacionados, que suele ser lo más difícil: averiguar, por ejemplo, si los pacientes coincidieron en el mismo lugar en los mismos tiempos, si tuvieron actividades en común, si comieron las mismas cosas o en los mismos lugares, etc. En el caso de Alemania se revisaron hasta las comandas de los restaurantes para saber qué habían comido los comensales enfermos y determinar a qué alimento estaban ligados los contagios. También y por primera vez se usaron las fotografías que algunos de éstos se tomaron en el restaurante para verificar el tipo de platillos que consumieron.
Una vez que se identifican las posibles fuentes, hay que cotejar in situ si efectivamente esas fuentes siguen existiendo. Cuando se trata de contaminación de alimentos, la fuente puede ser el alimento mismo o quienes lo manipulan.
Peligros de una asociación íntima
Dependiendo de sus mecanismos de infección y de los síntomas que producen, las E. coli patógenas se clasifican en ocho grandes grupos llamados patotipos. En particular, las E. coli enteroagregativas, que causan diarreas acuosas persistentes, se llaman así porque producen en su superficie una serie de "filamentos" (técnicamente fimbrias o pili) que les permiten unirse entre ellas y adherirse sorprendentemente bien a nuestras células intestinales, formando grandes agregados. De hecho, las E. coli de esta variedad se pegan mucho mejor a nuestros intestinos que las que causan usualmente las diarreas sanguinolentas y el síndrome urémico hemolítico (enterohemorrágicas). En otras pa labr a s, la nueva bacteria es un contrincante extremadamente eficaz: se pega fuertemente al interior de nuestros intestinos, por lo cual resulta muy difícil desalojarla de ahí, pero además produce una poderosa toxina que puede dañar de forma irreversible los riñones, causar una anemia muy grave y una horrible diarrea sanguinolenta en los enfermos. Peor todavía, si un paciente incauto toma antidiarreicos, lo único que logrará es que sus intestinos retengan durante más tiempo la toxina, con lo cual empeorará sensiblemente su afección; si se autorreceta antibióticos, puede agravar seriamente sus síntomas, ya que esta bacteria tiene la característica de que al percibir que algún antibiótico le está haciendo daño, libera aún más toxina.
Por fortuna, nuestro sistema inmunitario suele ser lo suficientemente efectivo como para contender con la dolencia en un máximo de 10 días. Sin embargo, las personas que llegan a desarrollar el síndrome urémico hemolítico necesariamente requerirán ser hospitalizadas, por varias razones: en primer lugar, para evitar que se deshidraten; en segundo, porque seguramente van a necesitar hemodiálisis para limpiar la sangre, ya que pueden tener dañados los riñones, que son los que suelen realizar esta función; en tercero, porque podrían requerir transfusiones sanguíneas a fin de corregir la anemia que se desarrolla durante la afección. En Alemania se está probando, en ocho pacientes, un medicamento experimental (Soliris) para evaluar los beneficios que pudiera tener en el combate a esta enfermedad. Sin embargo, dado su elevado precio, aún de tener éxito el fármaco no se aplicará a mayor escala, al menos en este momento.
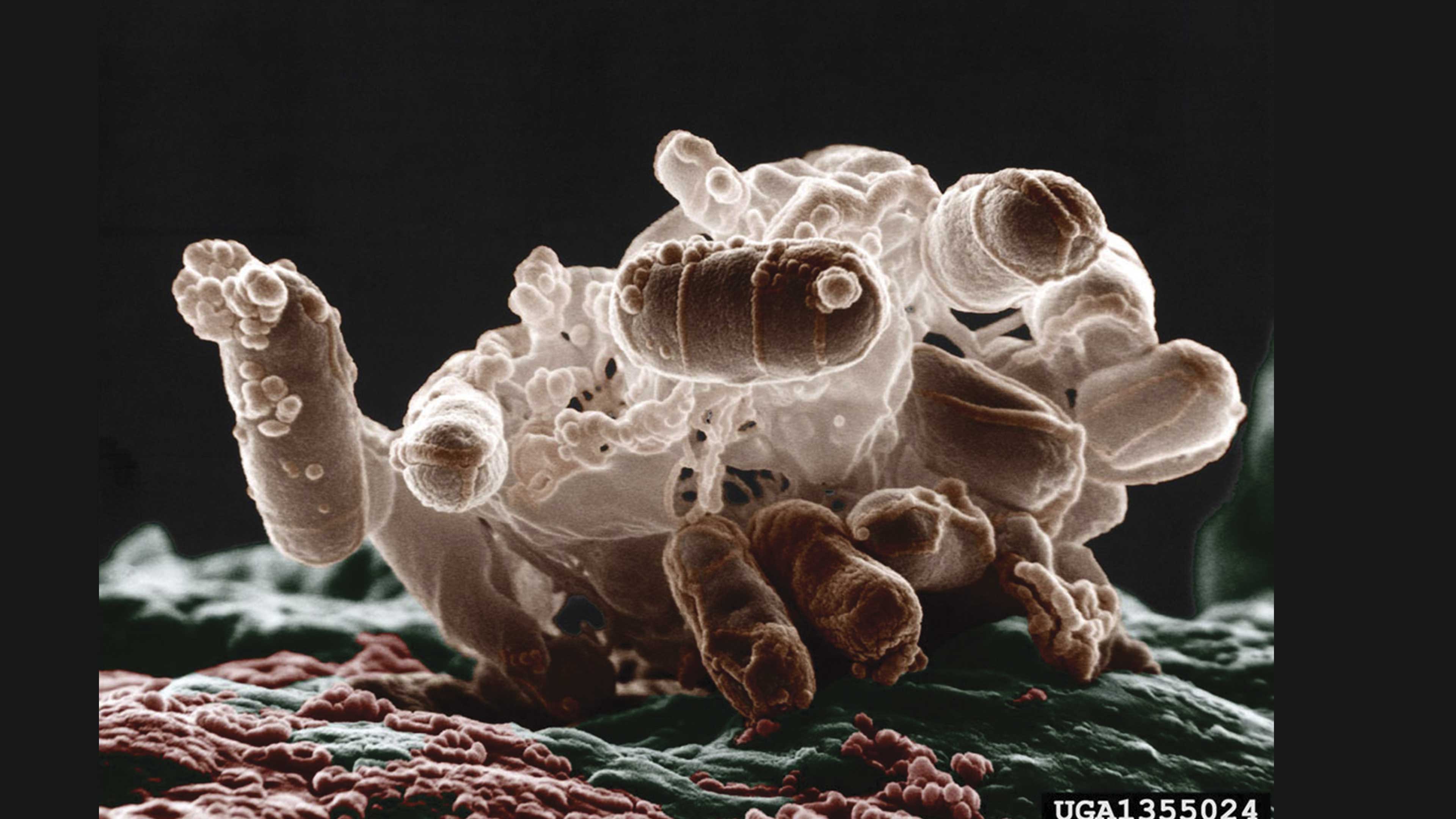 E. coli.
E. coli.
Un poco de historia
Aquellos que piensan que la E. coli es un organismo advenedizo en nuestros intestinos y que su presencia no es más que el resultado de los pecados de la vida moderna están completamente equivocados: los sesudos estudios de evolución molecular hechos por los investigadores estadounidenses Howard Ochman y Alex Wilson indican que la E. coli y sus parientes cercanos surgieron al mismo tiempo que los primeros mamíferos, hace entre 120 y 160 millones de años. Por primera vez tuvimos idea de la existencia de este pequeño organismo en 1886, cuando el p edia - tra austriaco Theodor Escherich lo aisló de heces fecales de niños sanos y lo describió bautizándolo como Bacterium coli commune. Después de la muerte de dicho pediatra, y en honor a su trabajo, la bacteria fue rebautizada con el nombre que hoy ostenta: Escherichia coli.
Desde que el ser humano existe, las Escherichia coli patógenas nos han provocado diarreas que han sido y siguen siendo en algunas partes del mundo una causa importante de mortalidad infantil. Sin embargo, fue hasta 1897 cuando por primera vez se sospechó que existían dos tipos de E.coli: las patógenas y las no patógenas, y tuvieron que pasar casi 50 años para que este hecho fuera confirmado gracias a las investigaciones del médico británico John S. B. Bray. En 1922, investigadores de la Universidad de Stanford hicieron una colección de E. coli aisladas de heces fecales humanas, sin saber que uno de sus miembros, la etiquetada como K12, se convertiría en el organismo mejor estudiado del planeta. El salto a la fama de E. coli K12 ocurrió en los años 40, cuando los investigadores Charles E. Clifton y Edward L. Tatum la eligieron para estudiar el metabolismo del nitrógeno y la síntesis del aminoácido triptofano. Pronto descubrieron que era muy fácil manipularla en el laboratorio, razón por la cual ésta se convirtió en el caballito de batalla de muchísimos investigadores, tanto así que en el transcurso de los 70 años siguientes sobre ella se han publicado cientos de miles de artículos científicos. En 1997, la E. coli K12 se convirtió en el sexto organismo de cuyo ADN (genoma) se obtuvo la secuencia completa, labor que le tomó seis años de arduo trabajo a un equipo conformado sobre todo por investigadores de la Universidad de Wisconsin, Estados Unidos.
La tecnología actual permite secuenciar genomas completos en unos cuantos días, por lo cual a la fecha se conoce el genoma de más de 1 500 bacterias, y entre ellas más de 50 E. coli, tanto no patógenas como de todos los patotipos. Una muestra palpable de que las técnicas para secuenciar ADN han mejorado notablemente, es el hecho de que la secuencia del ADN de la E. coli alemana se obtuvo en tan solo tres días, y a un costo muchísimo menor del que tuvo secuenciar el genoma de la E. coli K12.El análisis del material genético de la E. coli alemana arrojó muchas sorpresas: la primera, que ya mencionamos, el que esta bacteria, que al principio se pensaba que era una E. coli enterohemorrágica, resultara una E. coli enteroagregativa con la capacidad de producir verotoxina; la segunda, que contiene la información genética necesaria para producir otras 32 toxinas diferentes y al menos tres hemolisinas (proteínas que tienen la capacidad de romper los glóbulos rojos); otra más, que posee una gran variedad de genes que le confieren la capacidad de producir una enfermedad en el ser humano y que a grosso modo clasificamos como genes de virulencia. La E. coli alemana, además, posee tres paquetitos de información genética adicionales a la molécula de ADN que contiene la mayor parte de los genes de la bacteria y que se conoce como cromosoma. Dos de estos paquetitos, que técnicamente se denominan plásmidos, portan información relevante: uno de ellos tiene los genes que se requieren para fabricar las fimbrias que permiten a la bacteria pegarse firmemente a nuestras células intestinales y a otras superficies, mientras que el otro, el más grande, contiene los genes que le permiten vivir sin ninguna preocupación en presencia de 31 antibióticos diferentes.
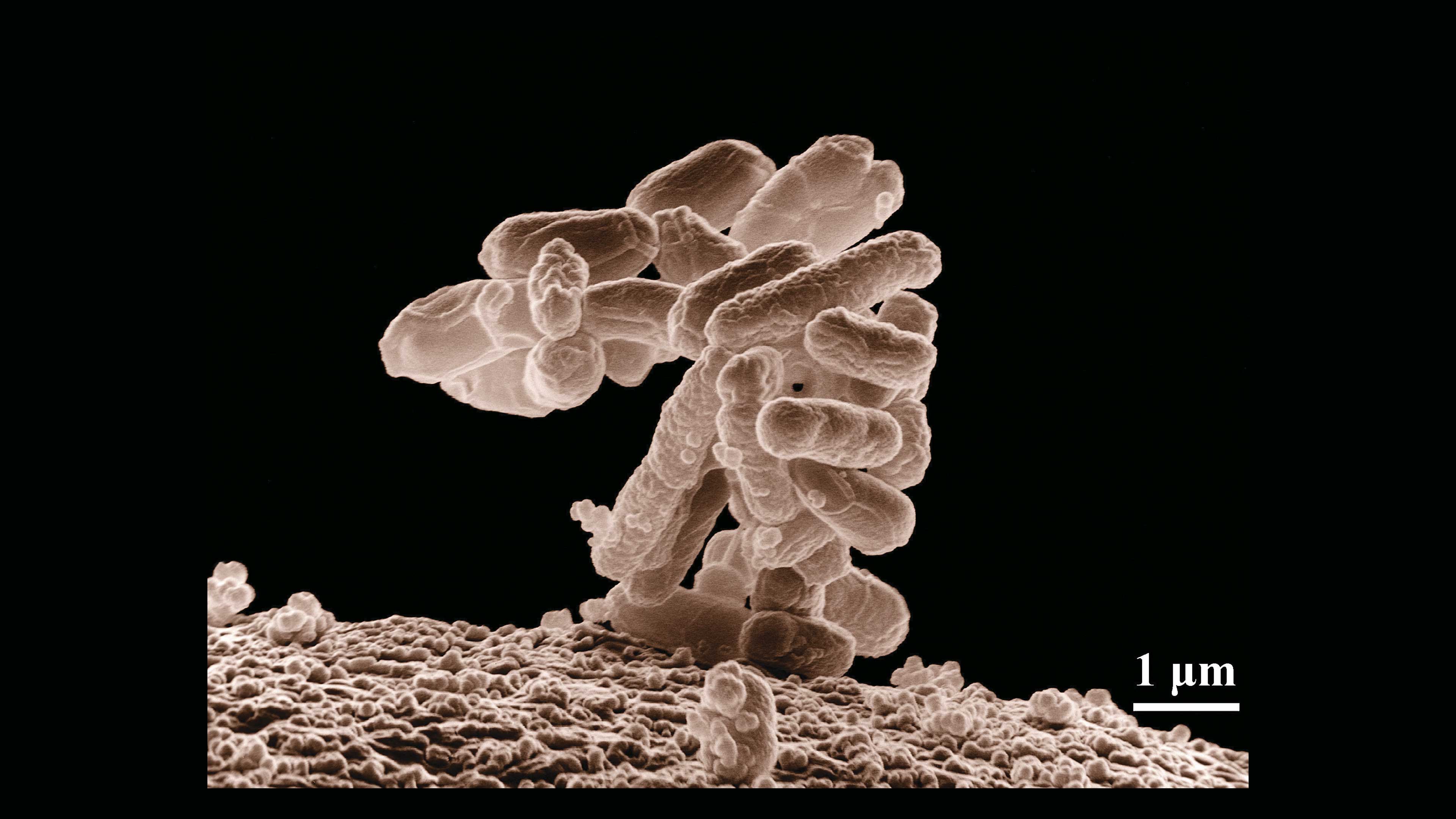 E. coli amplificada 10 000 veces.
E. coli amplificada 10 000 veces.
Promiscuidad, arma secreta de las bacterias
Uno de los descubrimientos más importantes en el campo de la microbiología, es que las bacterias son capaces de incorporar información genética de sus vecinos sin que importe (casi) la especie de que se trate. Si una bacteria se encuentra, por ejemplo, en un hospital, donde abundan los antibióticos, puede "tomar" los genes de un congénere resistente a estas sustancias y usarlos en su propio beneficio. Si necesita degradar petróleo para alimentarse y no cuenta con esa habilidad, puede adquirirla incorporando la información genética de una vecina que sí la posee. Si se encuentra en un ambiente donde las altas concentraciones de metales hacen difícil la vida, puede hacer suyos los genes que le confieran la capacidad de enfrentar esta agresión, consiguiéndolos de otra bacteria. En una perspectiva humana, sería como tener la posibilidad de tomar genes de un cactus para poder así contender con las sequías que se avecinan por el calentamiento global. Cuando se pudo contrastar el genoma de las E. coli patógenas con el de las no patógenas se hicieron descubrimientos que nos permiten entender cómo surgen las E. coli relacionadas con brotes epidémicos. El genoma de las E. coli patógenas es casi 25% más grande que el de las E. coli no patógenas. La información adicional se encuentra a manera de "parches" a lo largo del cromosoma bacteriano, y contiene muchos de los genes relacionados con la habilidad que posee la bacteria para generar una enfermedad, razón por la cual se les conoce como islas de patogenicidad. Otra observación es que estas islas se pueden encontrar en otras bacterias patógenas diferentes a E. coli, fuerte evidencia de que tienen la propiedad de transmitirse de una especie a otra sin problemas.
Se ha podido determinar que todas las E. coli tanto patógenas como no patógenas tienen en común alrededor de 2 200 genes; por ello a estos genes se les considera como el genoma medular (del inglés core genome) de esta especie. En contraste, los genes que no son comunes a todas las E. coli y que se supone que pueden ser todos transferidos de una bacteria a otra, son al menos 13 000. Los microbiólogos afectos a acuñar nuevos términos se refieren al conjunto de genes que tienen la capacidad de transferirse de un individuo a otro como el moviloma, nombre sin duda horrible, pero también sugerente. Queremos recalcar que en muchas ocasiones el moviloma puede compartirse entre especies bacterianas distintas. Otro término en boga es el pangenoma, es decir, el conjunto de genes que son patrimonio de la especie. En el caso de E. coli, su pangenoma consiste en los 2 200 genes del genoma medular, más los 13 000 del moviloma.
Cadena de eventos
A las E. coli, como a los demás organismos de esta Tierra, lo que más les interesa es seguir subsistiendo como especie, así es que adoptan cualquier estratagema que les permita tener más descendientes que los otros.
Cuando una E. coli patógena infecta a un individuo, no hace más que explotar un nuevo nicho ecológico para reproducirse en abundancia; las diarreas que provoca son una medida que utiliza la bacteria para dispersarse y permitir que sus descendientes encuentren otras víctimas a quienes infectar para repetir así nuevamente el ciclo. Cada vez que una E. coli obtenga genes del moviloma que le permitan infectar más eficientemente a los individuos, tendrá la potencialidad de reproducirse con pocas restricciones, al menos por un rato, de generar una epidemia y de fortalecer sus posibilidades de persistir como especie.
La E. coli alemana surgió como consecuencia de una cadena de eventos que seguramente se iniciaron cuando una E. coli enteroagregativa, capaz de generar diarreas fuertes, pero transitorias, incorporó genes de una E. coli enterohemorrágica. En otros dos eventos independientes adquirió dos plásmidos: uno de ellos le permitió resistir a muchos antibióticos; el otro, pegarse aún mejor en la superficie de nuestros intestinos. Sin duda, la adquisición adicional más importante fue el gen de la verotoxina, aportado por un virus bacteriano (bacteriófago) que puede fácilmente traspasar información genética de una bacteria a otra. Todos estos genes, parte del moviloma, se conjuntaron en la E. coli alemana para hacer de ella un patógeno muy efectivo, capaz de propagarse con rapidez. Afortunadamente, hoy contamos con programas muy eficientes de vigilancia epidemiológica y con estrategias que nos permiten mitigar con prontitud los brotes epidémicos.
El manejo de la epidemia en Alemania y de otras epidemias que han ocurrido recientemente (baste mencionar la de la influenza H1N1), nos muestran cómo la tecnología y la comunicación global permiten responder de manera coordinada y eficiente para identificar, caracterizar y delimitar un problema epidemiológico. Sin embargo, también nos revela muchas de nuestras limitaciones, así como los problemas económicos, políticos y sociales que suelen presentarse durante el manejo de los brotes epidémicos. Es también un buen ejemplo de cómo la naturaleza sigue su curso, y de cómo muchas veces la influencia humana lo altera.
El estudio de esta epidemia nos permite entender los mecanismos que utilizan las bacterias para renovar su información genética a fin de generar variantes con potencial epidémico. No nos queda sino permanecer siempre atentos y aprender de estas experiencias.
Más información
Miguel Ángel Cevallos, frecuente colaborador de ¿Cómo ves?, es doctor en investigación biomédica básica y especialista en genética molecular bacteriana. Trabaja en el Centro de Ciencias Genómicas de la UNAM.
José Luis Puente es investigador del Instituto de Biotecnología de la UNAM y experto en las Escherichia coli patógenas.














